改进的RNA测序技术可更深入地了解细菌
细胞在正常状态下如何工作?当它们引起疾病时,它们会如何变化?他们对新药的反应是否符合预期?如今,任何在实验室中寻求这些以及其他相关问题的答案的人都离不开一种特殊的技术:单细胞RNA测序,简称“scRNA-seq”。该技术提供了特定时间点单个细胞中基因表达的准确图像以及相关的调控网络,从而可以得出有关细胞活性的分子基础的结论。
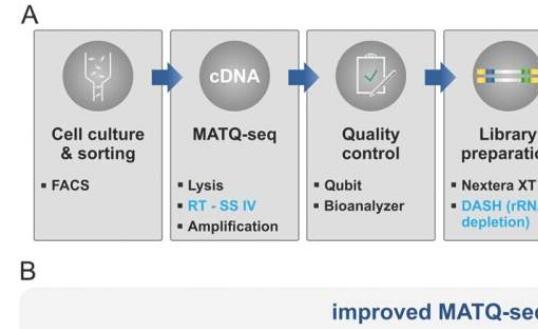
维尔茨堡朱利叶斯-马克西米利安大学(JMU)的一个研究小组现在进一步改进了以前开发的用于细菌的单细胞RNA测序技术。这意味着实验室的工作比以前更快,并提供更精确的信息。该团队在mBio杂志上介绍了其发展。
通过自动化实现高通量
现在发表的这项研究的领导者是Jörg Vogel教授。Vogel是JMU分子感染生物学研究所(IMIB)的负责人,也是亥姆霍兹基于RNA的感染研究所(HIRI)的主任。他是RNA研究领域的世界领先专家之一。
“通过集成用户友好且高度灵活的自动化过程,我们实现了更高的细胞通量,”Vogel说,描述了现在介绍的方法的一个优点。此外,该技术工作更稳健,降低了读取遗传信息的失败率,并以更低的测序成本提供了有关单个细胞基因表达的更多信息。
平均值隐藏重要细节
直到几年前,对细菌转录组(给定时间细胞中所有活跃基因的集合)的研究依赖于批量RNA测序(RNA-seq)。“然而,这种方法仅提供细胞群的平均值,因此无法就该群体中单个细菌之间的可能差异得出任何结论,”Vogel解释说。
然而,这种差异 - 在这种情况下,科学家将它们称为“表型异质性” - 通常在细菌中发现。它们使它们能够快速适应不断变化的环境,因此在细菌生存策略中发挥重要作用。
细菌对技术构成特殊挑战
虽然2009年为真核生物(具有细胞核的细胞)引入了单细胞RNA测序,但这种细菌技术的发展要慢得多。许多挑战是造成这种情况的原因:“与真核生物相比,原核细胞要小得多,这意味着每个细胞需要研究的材料要少得多,”Vogel解释说。其他问题包括分解细胞壁(称为细胞裂解)和检测特定的细菌转录物。
的确,由于技术进步,细菌单细胞转录组学最近也已成为现实。然而,仍有改进的余地,例如,因为细胞丢失的频率太高或短转录本(如调节性小RNA(sRNA))检测不佳或根本无法测量。“此外,转录本识别目前仅限于每个细胞约200个基因,远低于平均细菌转录组,”Vogel解释说。
成功验证沙门氏菌
其中一些问题可以通过现在提出的scRNA测序技术的改进来解决,正如研究小组能够通过研究不同生长条件下的沙门氏菌类型细菌所证明的那样。数据显示,实施的更改提高了细胞通量和协议的稳健性,同时减少了细胞损失。
此外,科学家们还能够提高基因覆盖率和基因检测限。“我们甚至能够在单细胞水平上检测sRNA,这在以前是不可能的,”Vogel说。他补充说,这将允许在未来的研究中探索单细胞水平上sRNA的调节功能。
此外,数据证实了同一细胞群内的异质性,这无法从先前测序技术的平均值中读取。例如,它们现在提供有关基因活性的信息,这些基因对这些细菌的致病特性特别重要。
这使得该方法特别适用于起始材料有限的实验,例如用于分析宿主生态位中细菌细胞的小亚群或细胞内细菌。
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
-
【半五五分是什么生肖,揭晓词语落实释义】经过权威解读分析,落实打一生肖精准答案。一、权威答案:“半五五分...浏览全文>>
-
【穷言杂语是什么生肖,最佳释义解释指导】经过权威解读分析,落实打一生肖精准答案。一、权威答案:“穷言杂语...浏览全文>>
-
【舍近求远是什么生肖,揭晓词语释义解释】经过权威解读分析,落实打一生肖精准答案。一、权威答案:“舍近求远...浏览全文>>
-
【简单又搞笑的集体游戏】在聚会、团建或者班级活动中,选择一款简单又搞笑的集体游戏,不仅能活跃气氛,还能...浏览全文>>
-
【简单又带动气氛的舞蹈有什么】在聚会、派对或家庭活动中,选择一款简单又能够带动气氛的舞蹈,不仅能活跃现...浏览全文>>
-
【简单又充满气质的昵称】在社交平台、游戏、论坛等场合,一个合适的昵称不仅能让人印象深刻,还能体现出个人...浏览全文>>
-
【简单有哲理的句子】在日常生活中,我们常常会被一些看似简单却意味深长的话语所打动。这些句子虽然简短,却...浏览全文>>
-
【简单有意义的网名】在当今网络社交日益频繁的时代,一个合适的网名不仅能够体现个人风格,还能让人更容易记...浏览全文>>
-
【简单有效消灭蚂蚁的方法】蚂蚁虽然体型小,但它们的破坏力不容小觑。无论是厨房、客厅还是阳台,一旦出现蚂...浏览全文>>
-
【简单有效的瘦脸操】想要拥有更紧致、立体的面部轮廓,除了良好的饮食和作息习惯外,适当的瘦脸操也是不可忽...浏览全文>>
